Technischer Tipp zur Probenvorbereitung
Niveau: Mittel
Peptid-Mapping-Probenvorbereitung - Gute Praktiken für tryptischen Verdau
Tipps in anderen Sprachen
Im Bereich von Biotherapeutika und Biosimilars stellen die Zulassungsbehörden Anforderungen an die Charakterisierung, die sich in erster Linie auf die physikochemischen Eigenschaften der Therapeutika konzentrieren, die die Wirksamkeit, Sicherheit und Immunogenität beeinflussen. Die Chromatographie spielt eine wichtige Rolle in drei spezifischen Bereichen der Analyse der physikochemischen Eigenschaften, nämlich der Struktur höherer Ordnung, der Primärstruktur und der posttranslationalen Modifikationen. Eine Schlüsseltechnik zur Bestimmung der Primärstruktur ist das Peptid-Mapping, bei dem in der Regel ein Serinprotease-Verdau eines gereinigten oder isolierten Proteins mit anschließender Analyse mittels HPLC durchgeführt wird. Die am häufigsten verwendete Protease für diese Art von Verdaus ist Trypsin und bereits innerhalb des Verdauschritts gibt es viele wichtige Praktiken, die wir zur Verbesserung des Ergebnisses anwenden können, welche im folgenden Technik-Tipp besprochen werden.
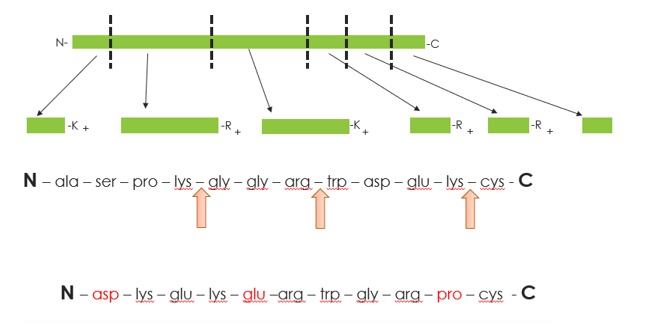
Die meisten Proteinverdaus werden aufgrund der gut definierten Spezifität mit Trypsin durchgeführt. Trypsin hydrolysiert nur Peptidbindungen, bei denen auf die Carbonylgruppe entweder ein Arginin- (Arg) oder Lysin- (Lys) Rest folgt, mit den folgenden Ausnahmen: wenn Lys oder Arg N-terminal an negativ geladene Aminosäuren gebunden sind oder wenn Lys oder Arg auf der C-terminalen Seite von Prolin (Pro) positioniert ist.
Trypsin wird zudem bevorzugt, da die resultierenden Peptide basische Reste am C-Terminus aufweisen, wodurch sie leichter durch Massenspektrometrie ionisiert werden können, was in der Regel die bevorzugte Nachweismethode für diese Anwendungen ist. Ein weiterer Vorteil der Verwendung von Trypsin ist der Massenbereich der resultierenden Peptide, der typischerweise zwischen 600 und 4000 Da liegt. Dies ist ideal für die anschließende Analyse, wenn entweder LC-UV oder LC-MS/MS verwendet wird.
Ein grundlegender tryptischer Verdau besteht aus einer Reihe von Schritten, für die im Folgenden einige Schlüsselpunkte des Prozesses zusammengefasst werden:
Denaturierung und Reduktion: Ein Chaotrop ist notwendig, um das Protein vollständig zu entfalten, so dass es für Trypsin zugänglich ist. Üblicherweise werden hierfür Harnstoff oder Guanidin verwendet. Es ist bekannt, dass Harnstoff die Sequenzabdeckung reduziert, da er zerfallen und anschließend Lysine und Cysteine carbamylieren kann, weshalb er in einigen Labors vermieden wird. Das am einfachsten zu verwendende Reagenz zur Denaturierung ist Guanidinhydrochlorid (GnHCl) welches jedoch vor dem Verdau mittels Pufferaustausch entfernt werden muss, da es die Trypsin Aktivität hemmt.
Alkylierung: Es gibt zwei übliche Alkylierungsmittel, Iodacetamid (IAM) und Iodessigsäure (IAA). IAM wird am häufigsten verwendet, da es schnell reagiert. Es wird jedoch auch IAA verwendet, da es weniger unspezifische Alkylierung aufweist. Bei beiden Reagenzien hilft auch die Zugabe von Dithiothreitol (DTT), eine weitere unspezifische Alkylierung zu verhindern.
Pufferaustausch: Nach der Alkylierung ist es entscheidend, die restlichen alkylierenden Reagenzien sowie alle denaturierenden Komponenten vor dem Verdau zu entfernen. Es gibt drei Hauptansätze für diese Art der Probenaufbereitung: Dialyse, Molekulargewichts-Cutoff-Spinfilter und Entsalzungssäulen. Die Dialyse führt zu langen Probenaufbereitungszeiten, weisst jedoch einen sehr geringen Probenverlust auf. Spinfilter und Entsalzungssäulen hingegen sind verlustanfällig, bieten aber eine schnellere Methode der Probenaufreinigung als die Dialyse.
Verdau: Hier betrachten wir Erwägungen zur Verdau mit Trypsin. Tryptische Peptide können relativ kurz sein und es besteht auch die Möglichkeit, dass Spaltungen ausgelassen werden. Schweinetrypsin wird am häufigsten für den Verdau verwendet; dieses Enzym hat eine Präferenz für Arg mit einer geringeren Effizienz für Lys. Außerdem findet keine Spaltung statt, wenn sich Asp oder Glu neben Lys oder Arg am C-Terminus oder Prolin (Pro) am N-Terminus befindet. Trypsin allein kann nur einen begrenzten Teil des Proteoms abdecken, wodurch parallel weitere Proteasen verwendet werden, darunter in vielen Fällen AspN oder GluC, um eine vollständige Sequenzabdeckung zu erreichen.
Wenn die Sequenzabdeckung kritisch ist, können orthogonale Proteasen verwendet werden, die im Folgenden zusammengefasst werden.
| Enzym | Spaltungsstellen | Vorteil | Einschränkugen |
|---|---|---|---|
| ArgC | C-Terminal von R | Proteom-Abdeckung qualitativ erhöhen | Größere Peptidfragmente als Trypsin |
| AspN | N-Terminal von D | Größer pH-Bereich | Größere Peptidfragmente als Trypsin. Verpasste Spaltungen sind möglich |
| Chymotrypsin | C-Terminal von F, Y, L, W | Orthogonaler Proteom Abdeckung zu Trypsin | Unpezifisch für hydrophobe Peptide, was zu verspassten Spaltungen führt |
| GluC | C-Terminal von D | Proteom-Abdeckung qualitativ erhöhen | Größere Peptidfragmente als Trypsin |
| LysC | C-Terminal von K | Ergänzt Trypsin | Überlappungen mit Trypsin, also keine ideale orthogonale Protease |
| Pepsin | C-Terminal von Y, F, W | Hohe Aktivität bei niedrigem pH-Wert für HDX-MS | Komplexe resultierende Spektren |
Weiter unten finden Sie eine Zusammenfassung von Parameter-Einschränkungen, die Sie bei dem Verdau von Proteinen mit Trypsin beachten sollten.
| Parameter | Bereich | Beispiel | Erwägungen |
|---|---|---|---|
| Verhältnis Enzym zu Substrat | 1:5 – 1:50 | 1:20 | Aufschlussdauer und -temperatur optimieren |
| Temperatur (°C) | 37-90 | 56 | Workflow-Durchsatz Deamidierung über 37 °C |
| Verdaudauer (Stunden) | 2-16 | 4 | Verdauvollsändigkeit Nichttrypsin-Aktivität, Trypsin-Autospaltung |
| pH | 7,5-8,2 | 7,6 | Deamidierung bei pH > 7,5 Optimale Trypsin-Aktivität |
| Ergänzende Protease | n/a | LysC | Vollständiger Verdau |
Zusammengefasst können wir sagen, dass es nicht immer die analytische Methode oder die HPLC-Analyse ist, die die Sequenzabdeckung vollständig bestimmt, wenn wir die Primärstruktur eines Proteins betrachten. Auch die Schritte und Vorsichtsmaßnahmen bei der Probenvorbereitung können eine wichtige Rolle spielen.
Einige davon sind oben zusammengefasst. Wenn bereits bei dem Verdau des Proteins geeignete Schritte unternommen werden, wird eine angenmessene Sequenzabdeckung in der anschließenden HPLC- oder LC-MS-Analyse um ein vielfaches einfacher zu erreichen.
ENTDECKEN SIE PhenoAcademy
PhenoAcademy ist ein KOSTENLOSES praktisches Schritt-für-Schritt-Webinar-Lernprogramm, das darauf ausgerichtet ist, Ihr Chromatographie-Wissen zu vertiefen und Ihre Forschungs- und Fehlerbehebungsfähigkeiten zu verbessern!



